Optimización del análisis de polimorfismos conformacionales de cadena simple. Caso gen del receptor de hormona Luteinizante bovino
Resumen
Con el objeto de optimizar el análisis de polimorfismos conformacionales de cadena simple (SSCP) para la detección y genotipado de los polimorfismos nucleotídicos simples (SNP) presentes en una región del gen receptor de hormona Luteinizante (LHR) bovino, se realizó un experimento en dos fases. En la primera se realizó un tamizaje molecular de 173 individuos mediante un análisis SSCP exploratorio. Las muestras que presentaron patrones electroforéticos diferenciales fueron secuenciadas y aquellas con variaciones nucleotídicas identificadas fueron utilizadas en la segunda fase para la optimización de la metodología SSCP. Se evaluaron las condiciones de la electroforesis variando la relación acrilamida/ bis-acrilamida, porcentaje de la mezcla, intensidad de corriente, porcentaje de glicerol y duración de la electroforesis. Los geles fueron teñidos en una solución de SYBER®safe, 0,7 %, durante 25 minutos en agitación suave y los patrones electroforéticos fueron visualizados mediante un transiluminador UV. La optimización de la técnica fue asumida basada en la separación, nitidez y perceptibilidad de las bandas, así como la detección de las mutaciones. Se consideró como optimizado el sistema electroforético I. Se confirmó la presencia del SNP rs41256848, además de la detección de una variante ubicada en la posición 1337 de la secuencia de referencia NM_174381.1. En esta investigación se optimizó un procedimiento práctico para la detección y genotipado de los polimorfi smos presentes en el fragmento de LHR bovino estudiado.
Descargas
Citas
• Ascoli, M., F. Fanelli and D. L. Segaloff. 2002. The lutropin/choriogonadotropin receptor, a 2002 perspective. Endocrine Reviews, 23(2), 141-174.
• Bastos, E., A. Cravador, J. Azevedo and H. Guedes-Pinto. 2001. Single strand conformation polymorphism (SSCP) detection in six genes in the Portuguese indigenous sheep breed “Churra da Terra Quente”. Biotechnology, agronomy, society and environment. 5(1), 7-15.
• Cerquera, F., O. Toro y G. Alberto. 2009. Frecuencias alélicas para variantes SNP en el gen Nramp1 en bovinos infectados con Brucella abortus o clasificados por resistencia al patógeno. Revista Corpoica (Colombia), 10, 43-50.
• Database of Single Nucleotide Polymorphisms (dbSNP). 2015. Bethesda (MD): National Center for Biotechnology Information, National Library of Medicine. dbSNP accession: {rs41256848-rs523043472}, (dbSNP Build ID: {126/143-142/143}). Available on line: https://bit.ly/33nUkGO [Mar. 24, 2015].
• Davis, J. 1994. Mechanisms of hormone action: luteinizing hormone receptors and second- messenger pathways. Current opinion in obstetrics gynecology. 6:254- 261.
• De La Rosa, O., A. Márques, B. Vásquez, G. Seijas y L. Dickson. 2013. Optimización de un protocolo para aislamiento de ADN a partir de sangre periférica en bovinos. Resumen presentado en el 2do Congreso Venezolano de Ciencia, Tecnología e Innovación. LOCTI - PEII, Caracas.)
• Dong, Y. and H. Zhu. 2005. Single-strand conformational polymorphism analysis: basic principles and routine practice. Methods in molecular medicine, 108, 149-157.
• Estrada-Cuzcano, A., J. Sandoval, M. Guevara- Fujita y R. Fujita. 2013. Uso de la técnica SSCP para detectar mutaciones puntuales del ADN mitocondrial humano. Revista Peruana de Biología, 12(3), 349-358.
• FAO. The State of the World’s Animal Genetic Resources for Food and Agriculture. 2007. Rome.
• Gasser, R., M. Hu, N. Chilton, B. Campbell, A. Jex, D. Otranto, C. Cafarchia, I. Beveridge and X. Zhu. 2007. Single-strand conformation polymorphism (SSCP) for the analysis of genetic variation. Nature Protocols, 1(6), 3121-3128.
• González, J., O. Saldarriaga, A. López- Herrera, N. Bermúdez, W. Zapata, J. Ossa, M. Rugeles and G. Bedoya. 2006. Polymorphism in 1908STR1934 locus of the 3 UTR of the Nramp1 bovine gene in eight cattle breeds. Revista Colombiana de Ciencias Pecuarias, 19(1), 11-17.
• Hastings, N., S. Donn, K. Derecka, A. Flint and J. Woolliams. 2006. Polymorphisms within the coding region of the bovine luteinizing hormone receptor gene and their association with fertility traits. Animal Genetics. 37(6): 583-585.
• Hayashi, K. 1991. PCR-SSCP: A simple and sensitive method for detection of mutations in the genomic DNA. PCR Methods Application Aug; 1(1):34-8.
• Huhtaniemi, I. 2000. The Parkes lecture. Mutations of gonadotrophin and gonadotrophin receptor genes: what do they teach us about reproductive physiology? Journal of reproduction and fertility. 119(2): 173-186.
• Humphries, S., S. Gudnason, V., N. Whittalland and M. Ian. 1997. Single-strand conformation polymorphism analysis with high throughput modifications, and its use in mutation detection in familial hypercholesterolemia. Clinical Chemistry 43:3, 427–435.
• Iniesta, R., E. Guino y V. Moreno. 2005. Análisis estadístico de polimorfismos genéticos en estudios epidemiológicos. GacSanit, 19(4): 333-341.
• Kakavas, V. K., P. Plageras, T. A. Vlachos and A. Papaioannou Noulas. 2008. PCR-SSCP: a method for the molecular analysis of genetic diseases. Molecular biotechnology, 38(2), 155-163.
• Kalvatchev, Z. and P. Draganov. 2005. Single- strand conformation polymorphism (SSCP) analysis: a rapid and sensitive method for detection of genetic diversity among virus population. Biotechnology & biotechnology equipment. 19/2005/3.
• Kim, E. S., X. Shi, O. Cobanoglu, K. Weigel,P. J. Bergerand and B. W. Kirkpatrick. 2009. Refined mapping of twinning-rate quantitative trait loci on bovine chromosome 5 and analysis of insulin-like growth factor-1 as a positional candidate gene. Journal of animal science, 87(3), 835-843.
• Korbie, D. and J. Mattick.2008. Touchdown PCR for increased specificity and sensitivity in PCR amplification. Nature protocols, 3(9): 1452 – 1456.
• Liefers, S., R. Veerkamp, M. te Pas, C. Delavaud, Chilliard, M. Platjeand and T. Lende. 2005. Leptin promoter mutations affect leptin levels and performance traits in dairy cows. Animal genetics, 36(2), 111-118.
• Marson, E., J. Ferraz, J.Meirelles, J. Balieiro and J. Eler. 2008. Effects of polymorphisms of LHR and FSHR genes on sexual precocity in a Bos taurus x Bos indicus beef composite population.
• Martínez, R., S. Dunner, R. Toro, J. Tobón,J. Gallego and J. Cañón. 2010. Effect of polymorphisms in the Slc11a1 coding region on resistance to brucellosis by macrophages in vitro and after challenge in two Bos breeds (Blanco Orejinegro and Zebu). Genetics and Molecular Biology, 33(3), 463-470.
• Martínez, R., R. Toro, F. Montoya, M. Burbano, J. Tobón, J. Gallego, S. Dunner and J. Cañón. 2008. Bovine SLC11A1 3′ UTR SSCP genotype evaluated by a macrophage in vitro killing assay employing a Brucella abortus strain. Journal of Animal Breeding and Genetics, 125(4), 271-279.
• Milazzotto, M. P., P. Rahal, M. Nichi, T. Miranda- Neto, L. A. Teixeira, J. B. S. Ferraz and J. F. Garcia. 2008. New molecular variants of hypothalamus–pituitary–gonad axis genes and their association with early puberty phenotype in Bos taurus indicus (Nellore). Livestock Science, 114(2), 274-279.
• Nataraj, A., I. Olivos-Glander, N. Kusukawa and W. Highsmith Jr. 1999. Single- strand conformation polymorphism and heteroduplex analysis for gel-based mutation detection. Electrophoresis, 20(6), 1177-1185.
• Till, B. J., T. Zerr, L. Coma and S. Henikoff. 2006. A protocol for TILLING and Ecotilling in plants and animals. Nature Protocols 1, 2465 - 2477.
• Orita, M., H. Iwahana, H. Kanazawa, K. Hayashi and T. Sekiya. 1989. Detection of polymorphisms of human DNA by gel electrophoresis as single-strand conformation polymorphisms. Proceedings of the National Academy of Sciences of the United States of America, 86(8), 2766- 2770.
• Paixão, T., C. Ferreira, A. Borges, D. Oliveira, A. Lage and R. Santos. 2006. Frequency of bovine Nramp1 (Slc11a1) alleles in Holstein and Zebu breeds. Veterinary Immunology and Immunopathology, 109(1-2), 37-42.
• Pazzola, M., M. Dettori, M. Atzeni, F. Balia and G. Vacca. 2010. Genetic diversity of NRAMP1 3’-UTR microsatellite in cattle breeds reared in Sardinia. Italian Journal of Animal Science, 8(2s), 126-128.
• Pruitt, K., G. Brown, S. Hiatt, F. Thibaud-Nissen, Astashyn, O. Ermolaeva, C. Farrell, J. Hart, M. Landrum, K. Mcgarvey, M. Murphy, N. O’Leary, S. Pujar, B. Rajput, S. Rangwala, Riddick, A. Shkeda, H. Sun, P. Tamez, R. Tully, C. Wallin, D. Webb, J. Weber, W. Wu, Dicuccio, P. Kitts, D. Maglott, T. Murphy and J. Ostell. 2013. RefSeq: an update on mammalian reference sequences. Nucleic acids research. 42: D756-D763. Avalaible on line: https://bit.ly/35zs2f4 [Ene. 01, 2014].
• Sherry, S., M. Ward, M. Kholodov, J. Baker, L. Phan, E. Smigielski and K. Sirotkin. 2001. dbSNP: the NCBI database of genetic variation. Nucleic acids research, 29(1):308- 311.
• Sunnucks, P., A. Wilson, L. Beheregaray, K. Zenger, J. French and A. Taylor. 2000. SSCP is not so difficult: the application and utility of single-stranded conformation polymorphism in evolutionary biology and molecular ecology. Molecular Ecology, 9(11), 1699–1710.
• Teschauer, W., T. Mussack, A. Braun, H. Waldner, and E. Fink.1996. Conditions for single strand conformation polymorphism (SSCP) analysis with broad applicability: a study on the effects of acrylamide, buffer and glycerol concentrations in SSCP analysis of exons of the p53 gene). European journal of clinical chemistry and clinical biochemistry: journal of the Forum of European Clinical Chemistry Societies, 34(2), 125-131.
• Walker, J., y R. Rapley. 2008. Molecular biomethods handbook (2nd ed ed.). Totowa, NJ: Humana Press.
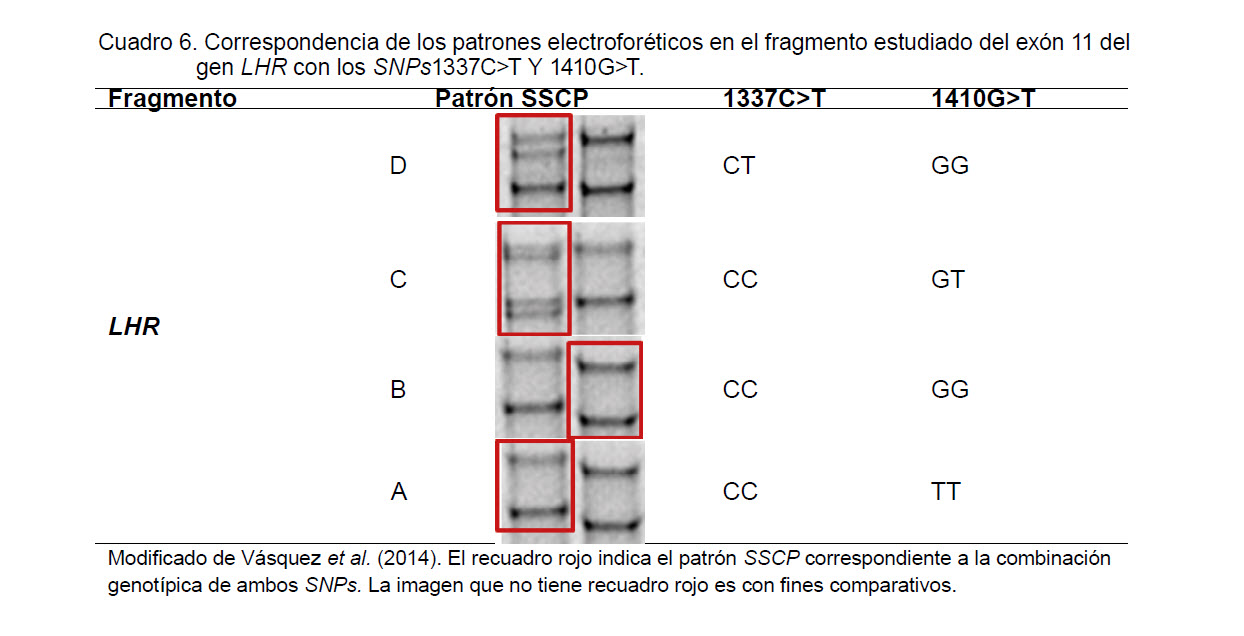
• Vásquez, B., A. Márques, G. Seijas, O. De La Rosa y J. Aranguren, J. 2014. Detección de polimorfismos en la región codificante del gen receptor de hormona luteinizante mediante análisis de polimorfismo conformacional de cadena simple y secuenciación en bovinos Carora. Revista Científica, FCV-LUZ / Vol. XXIV, Nº 5, 428-435, 2014
• Vignal, A., D. Milan, M. San Cristobal, and A. Eggen. 2002. A review on SNP and other types of molecular markers and their use in animal genetics. Genetic Selection Evol.34: 275-305.
• Yang, W. K. Tang, S. Li and L. Yang. 2011. Association analysis between variants in bovine progesterone receptor gene and superovulation traits in Chinese Holstein cows. Reproduction in domestic animals, 46(6), 1029-1034.
• Yu, Y., Y. Pang, H. Zhao, X. Xu, Z. Wu, L. An and J. Tian. 2012. Association of a missense mutation in the luteinizing hormone/choriogonadotropin receptor gene (LHCGR) with superovulation traits in Chinese Holstein heifers. Journal of Animal Science and Biotechnology, 3(1), 35.
Derechos de autor 2014 Belkys J. Vásquez M., Oscar De La Rosa, Alexis F. Márques U., Geomar Seijas Pedroza, José A. Aranguren Méndez

Esta obra está bajo licencia internacional Creative Commons Reconocimiento-NoComercial-CompartirIgual 4.0.








